SNP Pol DNA-Polymerase
Artikel-Nr.: M3009.0250
Shipping: Shipment on wet ice. Store at -20°C. For laboratory usage only!Sofort versandfertig, Lieferzeit 1-2 Werktage
*Preise zzgl. gesetzlicher MwSt. zzgl. Versandkosten
Infos Lieferbedingungen & Versandkosten >>
SNP Pol DNA-Polymerase für die einfache, verlässliche und schnelle spezifische Unterscheidung von Allelen, z. B. bei CRISPR/Cas9-gesetzten Punktmutationen, beim Erkennen falscher CRISPR/Cas9-Produkte oder bei der Validierung von Sequenzierergebnissen. Die SNP Pol DNA-Polymerase erkennt hochspezifisch, ob eine Fehlpaarung (Mismatch) des Primer-Template-Komplexes vorliegt oder nicht. Die Fehlpaarung (Punktmutation) muss sich am 3´-Ende des Primers befinden. Somit können mutante Allele exakt von Wildtypallelen unterschieden werden - ohne Sequenzierung, da die Polymerase im Fall einer Fehlpaarung schlichtweg nicht amplifiziert.
Legen Sie Ihren Primer einfach auf die angenommene Punktmutation (Wichtig: Die Punktmutation muss am 3'-Ende sein) und die Polymerase erkennt eine Fehlpaarung in diesem Bereich mit 100% Genauigkeit: Wenn die zum 3'-Ende des Primers komplementäre Base auf dem DNA-Strang die Mutation zeigt und der Primer nicht, dann erfolgt keinerlei Amplifikation - schnelle Gewissheit zu 100%!
Die SNP Pol DNA-Polymerase kann daher einfach, zeit- und kosteneffizient für das Screening von Punktmutationen eingesetzt werden.
Die Variante SNP PolTaq DNA-Polymerase > besitzt 5'-3'-Nukleaseaktivität und kann daher mit spezifischen Primersonden wie Taqman® Sonden oder Molecular beacons eingesetzt werden.
Einmaliges Testmuster zum Sonderpreis erhältlich. Keine Versandkosten bei Versand innerhalb Deutschlands! Der Testmusterpreis wird bei der ersten offiziellen Bestellung des Produkts zurückerstattet.
Beschreibung
Die SNP Pol DNA-Polymerase ist eine hochselektive DNA-Polymerase zur Detektion von Single Nucleotide Polymorphisms. Sie wurde speziell für die allelspezifische Diskriminierung entwickelt, bei der eine sehr hohe Unterscheidungsrate (high discrimination) benötigt wird: z.B. bei der Allelspezifischen PCR (ASA; AS-PCR), Allelspezifischen Primerextension (AS-PEX), SNP Analyse, Genotypisierung oder bei methylierungsspezifischen PCRs (MSP). Viele DNA-Polymerasen tolerieren fehlgepaarte Primer-Template Komplexe. Die SNP Pol DNA-Polymerase dagegen unterscheidet diese spezifisch (high discrimination) und liefert gezielt nur PCR-Produkte bei perfekt passenden Primerpaaren! Die Genaxxon SNP Pol und SNP PolTaq DNA-Polymerasen unterscheiden bis zu 100% mittels allelspezifischer PCR zwischen den beiden Allelen und ergeben nach einer simplen qPCR eine eindeutige Aussage, welches Allel vorliegt. Somit kann das prinzipiell große Potential der CRISPR/Cas9 Technologie mit der SNP PolTaq bzw. der SNP DNA-Polymerase für die Humanmedizin und Pflanzenbiotechnologie genutzt werden.
Mittels allelspezifischer PCR kann man die Mutationsrate in einem Pool oder Hintergrund von Wildtyp-Sequenzen quantifizieren. Auch die Überprüfung von Mutationshäufigkeiten, die durch NGS bestimmt wurden, kann mittels allelspezifischer PCR und SNP Pol DNA-Polymerase verifiziert werden. Sehr gut geeignet ist die SNP PolTaq DNA-Polymerase zur Analyse von Liquid Biopsie Proben. Mit ihr kann die Anwesenheit und Häufigkeit von Krebsmutationen sehr gut analysiert und quantifiziert werden.
Bild: Anwendungsbeispiel SNP Pol DNA Polymerase
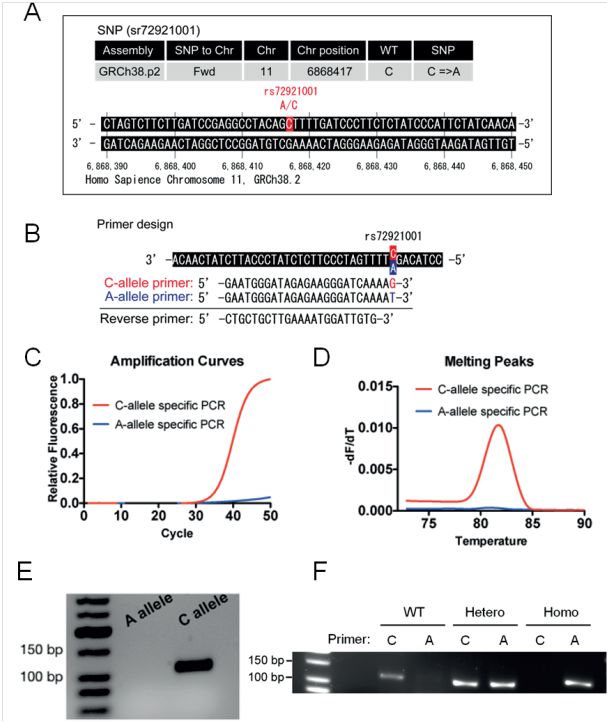
Wir empfehlen die Anwendung von Primern für kurze Amplicons (ca. 60-200 bp) für optimale Ergebnisse. Längere Amplicons sind ebenfalls möglich.
Bei längeren Amplicons >500 bp ist unter Umständen der Zusatz von Magnesium (+0.5 - 1.5mM) erforderlich.
Trouble shooting:
Keine Banden nach PCR von 30 Zyklen!
Optimierungsvorgehen bei fehlender oder schwacher Bande
- realtime PCR Ansatz
- Überprüfen der dNTP-Konzentration. Diese sollte zwischen 200 und 300µM (in der PCR) liegen.
- Erhöhen der Zyklenzahl (mindestens 35, besser gleich 40)
Test Optimierung - Zyklenzahl
Bei der Endpunktsdetektion ist eine Zyklenanzahl von 30 nicht immer ausreichen, um eine gut sichtbare Bande zu generien. Zum Beispiel bei weniger als 200 DNA-Kopien als Ausgangswert. Daher sollte der Test mittels realtime PCR gefahren werden, oder man setzt fünf parallele PCRs an, von denen jeweils eine nach fünf unterschiedlichen Zyklen aus dem PCR-Gerät entnommen wird (Beispiel unten).
Endpunktsdetektion:
Setzen Sie parallel fünf gleiche PCR-Reaktionen mit der SNP Pol DNA-Polymerase an.
Nehmen Sie bei 20, 25, 30,35 und 40 Zyklen jeweils eines der PCR-Tubes aus dem Cycler und tragen am Ende alle fünf Reaktionen auf ein Agarosegel auf.
Damit lässt sich dann einfach erkennen, welches die optimale Zyklenzahl in der PCR für die gegebene Anwendung (Ausgangsmaterial, Target, Primer) ist.
SNP Pol PCR mit Zelllysaten
Tierische Zellen und E.coli können direkt für die PCR mit der SNP Pol DNA-Polymerase herangezogen werden! Es ist keine separate Lyse und kein Proteinase K-Verdau notwendig.
Vorgehen PCR-Ansatz:
1. Es werden 50 - 500 Zellen pro PCR-Ansatz benötigt!
2. PCR Mix mit Primern und Puffer vorbereiten und auf Eis stellen!
3. Die gepickte Kolonie direkt in 10-20µL Wasser (keine separate Lyse und kein Proteinase K-Verdau) geben,
kurz vortexen (5 Sekunden), diese Zell-Suspension dann direkt in den PCR-Reaktionsansatz geben, z.B.
10µL Zellsuspension für einen PCR-Ansatz mit 20µL Gesamtvolumen. Anfängliche Denaturierungszeit von 2-3 Minuten
reicht aus, kann notfalls auf 5 Minuten verlängert werden.
Genomische DNA pro Reaktion max. 100 Kopien ist relativ gering, sollte aber noch gehen.
Der Rest dieser Zell-Suspension kann auch weggefroren werden, um weitere Tests damit durchzuführen.
Optional:
4. wenn es möglich ist: real-time PCR machen!
Die SNP Pol DNA-Polymerase (M3009 >) oder (M3061 >) kann zusammen mit unspezifischen Fluoreszenzfarbstoffen (z.B. Green DNA Dye > von Genaxxon oder SybrGreen®) in der realtime PCR eingesetzt werden. Wenn mit spezifischen PCR-Probes gearbeitet wird, ist nur die SNP PolTaq DNA-Polymerase dafür einsetzbar, da nur diese eine 5'-3' Exonukleaseaktivität besitzt.
Mit unseren hochwertigen dNTPs als Set (M3015.4100 und M3015.0250) > oder als Mix (M3016.1010) > oder unseren DNA-Markern > und unserer günstigen Standardagarose > bieten wir Ihnen weitere Produkte für die PCR.
Anwendungsgebiete für die SNP Pol DNA-Polymerase
- Monitoring, verification and detection of point mutations
- Identification of correct or wrong CRISPR/Cas9 products
- Verification/validation of sequencing results
- Quantification of mutations (e.g. NGS results)
- SNP-detection by allele-specific amplification (ASA) / Allele-specific PCR
- Methylation specific PCRs (MSP) after bisulfite treated DNA (CpG methylation sides)
- HLA genotyping
- micro sequencing
- realtime PCR with hydrolysis probes
- realtime multiplex PCRs
- DamID-seq data in C. elegans.
Sharma R, Ritler D, Meister P.
Tools for DNA adenine methyltransferase identification analysis of nuclear organization during C. elegans development. Genesis. 2016 Feb 4. doi: 10.1002/dvg.22925.
- Minisequencing SNP genotyping with SNPase DNA Polymerase can be carried out by the procedure described in:
Lovmar L, Fredriksson M, Liljedahl U, Sigurdsson S, Syvänen AC.
Quantitative evaluation by minisequencing and microarrays reveals accurate multiplexed SNP genotyping of whole genome amplified DNA. Nucleic Acids Res. 2003;31:e129.
- Allel specific mismatch selectivity by the HiDi DNA polymerase
high single nucleotide discrimination
Technische Daten:
Specifications:
SNP Pol DNA polymerase is supplied as a 5 U/µL solution. It comes together with an optimized 10x reaction buffer.
SNP Pol DNA polymerase shows no 5´-3´ exonuclease activity! Not applicable for usage together with hydrolysis probes for, e.g., TaqMan™ probes.
Applikation:
- Monitoring, verification and detection of point mutations - Identification of correct or wrong CRISPR/Cas9 products - Verification/validation of sequencing results - Quantification of mutations (e.g. NGS results) - SNP-detection by allele-specific amplification (ASA) / Allele-specific PCR - Methylation specific PCRs (MSP) after bisulfite treated DNA - HLA genotyping - micro sequencing - realtime PCR (without hydrolysis probes; use SNP PolTaq) - realtime multiplex PCRsQuelle
rec. from E.coliSicherheits Hinweise / Safety
Klassifizierungen / Classification
eclass-Nr: 32-16-05-02Dokumente - Protokolle - Downloads
Hier finden Sie Informationen und weiterführende Literatur zu SNP Pol DNA-Polymerase. Für weitere Dokumente (Zertifikate mit weiteren Lotnummern, Sicherheitsdatenblätter in anderer Sprache, weitere Produktinformationen) wenden Sie sich bitte an Genaxxon biosience unter: info@genaxxon.com oder Tel.: +49 731 3608 123.
Dokumente - Protokolle - Downloads
Kann man die SNP Pol DNA-Polymerase mit Bisulfit-behandelten DNA-Proben verwenden?
Ja, unsere SNP Pol DNA-Polymerase ist beispielsweise hervorragend geeignet für die methylierungsspezifische PCR (MSP). Da eine einzelne Fehlpaarung für bestimmte MSP-Ergebnisse ausreicht, ermöglicht SNP Pol DNA-Polymerase sogar eine zuverlässige MSP-Analyse einzelner CpG-Stellen.
Kann man SNP Pol 2x PCR Master Mix und SNP Pol DNA-Polymerase in auf Sonden basierenden Assays verwenden?
Ja, das ist möglich, solange keine Nukleaseaktivität erforderlich ist. Bitte beachten Sie, dass SNP Pol DNA-Polymerase keine 5'-3'-Nuklease-Aktivität aufweist. SNP Pol 2x-PCR Master Mix kann nicht in hydrolysebasierten Sondenassays (z. B. TaqMan®) verwendet werden. Für diese Assays empfehlen wir die Verwendung unserer SNP PolTaq DNA-Polymerase mit 5'-3'-Nuklease-Aktivität. Die SNP PolTaq-Variante kann daher für auf Hydrolysesonden-basierenden Real Time-PCRs verwendet werden.
Was ist die Fehlerrate von SNP Pol DNA-Polymerase?
Die Fehlerrate von SNP Pol ist vergleichbar mit der Fehlerrate von Wildtyp-Taq DNA-Polymerase.
Kann man SNP Pol DNA-Polymerase in Real Time-PCRs mit einem qPCR-Farbstoff wie SYBR® Green verwenden?
Bitte beachten Sie, dass SNP PolTaq DNA-Polymerase nicht für Real Time-PCR mit einem qPCR-Farbstoff geeignet ist. SNP Pol DNA-Polymerase funktioniert sehr gut mit qPCR-Farbstoffen.
Für welche Amplikonlänge sollten Primer ausgelegt werden?
Wir empfehlen, Primer mit einer kurzen Amplikonlänge (ca. 60-200 bp) zu entwerfen, um optimale Ergebnisse zu erzielen. Es sind jedoch auch längere Amplikonlängen möglich.